Rede Corona-Ômica BR-MCTI divulgou informe com resultado de análises realizadas no Laboratório de Microbiologia Molecular da Feevale
Nova análise divulgada em informe da Rede Corona-Ômica BR-MCTI, integrante da Rede Vírus do Ministério da Ciência, Tecnologia e Inovações (MCTI), revelou que a variante Delta (21J) do SARS-CoV-2, vírus causador da Covid-19, já representa 100% das amostras analisadas em 10 cidades* do Rio Grande do Sul. Por meio do Laboratório de Microbiologia Molecular da Universidade Feevale, foram identificados e sequenciados 91 genomas completos de SARS-CoV-2 de amostras coletadas em municípios das regiões Metropolitana, Vale do Sinos, Serra e Caí, entre os dias 29 de outubro e 7 de dezembro.
Detectada pela primeira vez em junho, a variante Delta se mostrou predominante nos sequenciamentos realizados pelo laboratório desde agosto e sua frequência no último período analisado foi de 100%. Esse acompanhamento permitiu observar que a Gamma (P.1 e suas ramificações), variante predominante até julho e que gerou o maior impacto no Brasil e no RS no primeiro semestre de 2021, foi totalmente substituída pela variante Delta no Estado.
Com relação à caracterização genética, as sequências foram alinhadas com genomas completos de SARS-CoV-2 de diferentes variantes através da plataforma on-line NextClade e a árvore filogenética foi inferida. Todas as amostras foram classificadas como pertencentes ao clado 21J da variante Delta. Posteriormente, as sequências foram analisadas por meio da plataforma on-line Pangolin, onde foram caracterizadas como pertencentes às seguintes sublinhagens de Delta: AY.99.2 (60/91 – 65,94%), AY.101 (24/91 – 26,4%), AY.43 (4/91 – 4,4%) e AY.102 (1/91 – 1,09%), AY.34.1 (1/91 – 1,09%), AY.122 (1/91 – 1,09%).
De acordo com o pró-reitor de Pesquisa, Pós-graduação e Extensão da Universidade Feevale e coordenador da Rede Corona-Ômica BR-MCTI, Fernando Spilki, o grupo permanece vigilante e utilizando ferramentas de genotipagem e sequenciamento.
Assim, buscamos auxiliar na detecção e acompanhamento da futura dispersão da variante Ômicron no Brasil”, afirma Spilki.
*Amostras provenientes de: Campo Bom (24), Canoas (22), Estância Velha (6), Esteio (1), Garibaldi (1), Novo Hamburgo (30), Porto Alegre (3), São Sebastião do Caí (2), Sapucaia do Sul (1), Taquara (1). Os genomas obtidos no presente estudo devem ser depositados em bases nacionais e internacionais nos próximos dias.
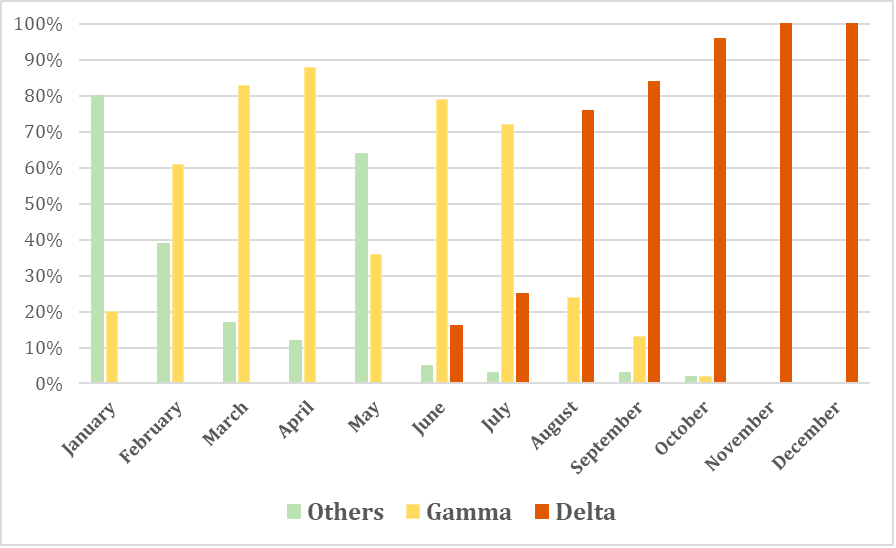
No gráfico ao lado: distribuição das linhagens de SARS-CoV-2 sequenciadas pelo Laboratório de Microbiologia Molecular na Universidade Feevale entre os meses de janeiro e dezembro de 2021.







